Freitag, März 06, 2009
Ich schmeiß hin...
Ab sofort gibts Neues von mir auf ScienceBlogs.de, bei "Alles was lebt". Aber nicht nur von mir, Ele von Selective Sweep ist auch an Bord!
Ich würde mich freuen, wenn ihr mir auch auf dem neuen Blog treu bleibt!
Achja, einen Feed gibt es auch schon.
Mittwoch, März 04, 2009
Wissenschaftliche Gründe, warum die EU "Genmais" verbietet: wohl keine...
Ich will jetzt keine Grundsatzdiskussion anwerfen, welche Gründe denn für oder gegen gentechnisch veränderte Organismen und deren Verwendung in der Landwirtschaft sprechen. Es geht mir eigentlich nur darum, welche ganz anderen Gründe unser Umweltminister Gabriel für seine Entscheidung bei der Abstimmung der EU-Umweltminister genannt hat. Es waren jedenfalls keine Umweltbedenken.
Ich kann nicht erkennen, warum wir den Interessen eines einzigen amerikanischen Konzerns folgen und in den Mitgliedstaaten die Bürger gegen uns aufbringen sollen[...]Die Leute wollen keine GVOs, also verbieten wir sie? Hoffentlich kriegen nicht zu viele Steuerzahler mit, wie Politik funktioniert...
Und was heißt, den Interessen eines einzigen amerikanischen Konzerns folgen? Da nur diese eine Maissorte europaweit die einzige zugelassene gentechnisch veränderte Nutzpflanze ist, hat das hier nichts mit einer Bevorzugung zu tun - auch die Konkurrenz von Monsanto kann GVOs erzeugen und dann von der EU für den Anbau erlauben lassen. Ber der negativen politischen Stimmung wird das nur keiner machen. Und wenn doch so viele Bürger gegen grüne Gentechnik ist, dann dürfte sich das doch auf dem Markt von selbst regeln, oder?
Quelle: biotechnologie.de
Dienstag, März 03, 2009
Die Transkription, animiert mit Lego
[via BoingBoing Gadgets]
Montag, März 02, 2009
Projekt Paperübersicht Intermezzo: Homologe Rekombination
Rekombination bedeutet zunächst, dass genetische Information zwischen zwei DNA-Molekülen ausgetauscht wird. Von den vielen Rekombinationsmechanismen ist die homologe Rekombination der konservativste. Das war jetzt keine politische Aussage, es bedeutet nur, dass es dabei idealerweise zu keiner Änderung der Sequenz kommt. Dies ist möglich, weil für die homologe Rekombination identische (= homologe) DNA-Sequenzen verwendet werden. Diese Sequenzen kommen bei diploiden Organismen wie dem Menschen oder Arabidopsis vom homologen Chromosom. Oder, noch besser, nach der Verdopplung der Chromosomen während der Replikation, vom Schwesterchromatid. Ausgangspunkt der homologen Rekombination ist immer ein Doppelstrangbruch. Dieser kann gewollt sein, etwa in der Meiose (dazu in einem anderen Teil der Paperübersicht mehr, wahrscheinlich #5). Alternativ kann ein Doppelstrangbruch aber auch durch ionisierende Strahlung oder Chemikalien ausgelöst werden. Dies wäre dann die Verbindung der homologen Rekombination mit der DNA-Reparatur.
Was wirklich während der homologen Rekombination passiert, wissen wir noch nicht hundertprozentig. Man kann diesen vorgang nicht live an einem DNA-Molekül mitverfolgen. Man kann aber seine Versuche so aufbauen, dass bestimmte Abläufe aufgrund des Ergebnisses ausgeschlossen werden können, andere Abläufe aber wahrscheinlicher werden. Viele solcher grundlegenden Experimente wurden mit der Bäckerhefe Saccharomyces cerevisiae gemacht, bei der clever definierte Konstrukte in das Genom eingebracht wurden, die nach einer homologen Rekombination erlauben, Anteile von bestimmten Vorgängen zu ermitteln. Das hört sich jetzt sehr nichtssagend an, deshalb will ich kurz an einem schönen Beispiel zeigen was ich damit meine.
Das "Double Strand Break Repair" Modell der homologen Rekombination
1983 fassten Szostak et al. in einem Review-Artikel den damals aktuellen Stand der Rekombinationsforschung zusammen. Dabei stellten sie fest, dass bisher vorgeschlagene Modelle der homologen Rekombination selten auftrende Ereignisse während der Meiose nicht vorhersagen konnten. Wenn man beispielsweise zwei Marker betrachtet [1], die heterozygot und gekoppelt vorliegen (also gemeinsam auf einem Chromosom sitzen, aber nicht auf dem zweiten Chromosom eines diploiden Organismus), dann erwartet man in den Nachkommen die einfache Aufspaltung nach Mendel in 1:1 - eine Hälfte der Nachkommen hat das Chromosom mit beiden Markern erhalten, die andere Hälfte das homologe Chromosom ohne die Marker. Aufgrund der besonderen Chromosomensituation der damals untersuchten Pilzarten ist die Notation hier traditionellerweise nicht 1:1, sondern 4:4, das ändert am Verhältnis aber nichts. Man kann nun aber beobachten, dass manchmal auch andere Aufteilungen passieren, beispielsweise 6:2 (bzw. 2:6), oder auch 5:3. Hier muss Information von einem Chromosom auf ein anderes übertragen worden sein, oder ein Austausch zwischen zwei Chromosomen erfolgt sein, so dass die beiden Marker nicht mehr gekoppelt auf einem Chromosom vorliegen und unabhängig voneinander vererbt werden können. Diese beiden Vorgänge bezeichnet man als gene conversion und crossing over, und sie sind in Abbildung 1 aus Szostak et al. dargestellt (wenn auch in umgekehrter Reihenfolge):

Abbildung 1 aus Szostak et al. (1983). Zustand von zwei gekoppelten heterozygoten Markern A und B und die Effekte von crossing over (a) und gene conversion (b). Klicken für größere Version.
Woran liegt das nun? Szostak et al. schlagen einen Mechanismus vor, der auf der Reparatur von Doppelstrangbrüchen beruht. Dies war neu, denn die bisherigen Modelle gingen von Auslösern wie kurzen einzelsträngigen Bereichen auf der DNA (ssDNA nicks) aus. Von der Reparaturforschung war damals bereits bekannt, dass die Doppelstrangbruchreparatur in der Bäckerhefe sehr effizient ablief. Das neue Modell sollte also mit Doppelstrangbrüchen beginnen, und mit der Möglichkeit für sowohl crossing over, als auch gene conversion enden. Die Idee von Szostak und Kollegen sah folgendermaßen aus:
Ausgehend von einem Doppelstrangbruch werden die freien Enden durch Proteine (Exonukleasen) so zurückgeschnitten, dass freie einzelsträngige Überhänge vorliegen. So eine ssDNA ist dann natürlich verfügbar für Basenpaarungen. Wenn nach einer "Homologiesuche" eine homologe DNA-Sequenz gefunden wird (die wie gesagt beispielsweise auf dem homologen Chromosom, oder auch dem Schwesterchromatid liegen kann), dann erfolgt die sogenannte Einzelstranginvasion: Der Einzelstrang lagert sich an die komplementäre Sequenz an und verdrängt dabei einen der vorhandenen Stränge. Die daraus resultierende Struktur wird D-Loop (displacement loop) genannt. Von hier an kann das freie Ende mit Hilfe einer DNA-Polymerase verlängert werden, was den D-Loop vergrößert. Irgendwann ist ein so großer Bereich DNA im D-Loop verdrängt, dass dieser mit dem zweiten freien Ende des Doppelstrangbruches paaren kann. Dies bedeutet auch, dass das erste freie Ende endlich mit der anderen Seite des Doppelstrangbruches verknüpft werden kann. Der DSB ist jetzt zwar repariert, aber wir haben nun eine problematische DNA-Struktur vorliegen - zwei DNA-Moleküle sind an zwei Positionen überkreuzt. So eine kreuzförmige DNA-Struktur nennt man übrigens nach ihrem Erstbeschreiber Holliday Junction (AHA!), bei den zwei Überkreuzungen hier spricht man von einer doppelten Holliday Junction (dHJ). Warum ist diese Struktur problematisch? Weil eine Zelle sich nicht teilen kann, bevor die dHJ aufgelöst wurde!
Und jetzt greift die Idee von Jack Szostak und Kollegen. Eine Endonuklease, also ein Protein das im Inneren eines DNA-Moleküles schneidet, kann an den Holliday Junctions Schnitte setzen, um die zwei Stränge voneinander zu trennen. Und abhängig davon, ob die beiden Schnitte symmetrisch (unten links) oder asymmetrisch (unten rechts) erfolgen, erhält man entweder ein crossing over (CO, rechts) oder eine gene conversion (auch noncrossover genannt, also NCO, links).
Seit 1983 ist viel Zeit vergangen, doch das DSBR-Modell hat sich gehalten. Mittlerweile wurde etwa gezeigt, dass in der Meiose ein spezielles Protein absichtlich Doppelstrangbrüche zur Einleitung der homologen Rekombination setzt: SPO11. Auch Holliday Junctions wurden bereits als Intermediate der Rekombination experimentell nachgewiesen.
"Synthesis-dependent strand-annealing" und das "revised model"
Um es jetzt noch ein wenig komplizierter zu machen, will ich der Vollständigkeit halber das Bild auf den aktuellen Stand bringen. Als 1994 die Gruppen von William Engels und Gregory Gloor (Nassif et al., 1994) Rekombinationsexperimente mit der Fruchtfliege Drosophila melanogaster machten, stießen sie auf Ergebnisse, die sich mit dem DSBR-Modell von Szostak et al. nicht vollständig erklären ließen. Letzen Endes waren ihre Ergebnisse nur zu verstehen, wenn man beiden freien Enden des DSB erlaubte, unabhängig voneinander die Rekombination mit verschiedenen Partnern einzuleiten. Eine Auflösung nach ihrem synthesis-dependent strand-annealing-Modell (SDSA) benötigte demnach keine Holliday Junction als Intermediat. Bereits nach Verlängerung des ersten freien Endes würde dieses aus dem D-Loop geworfen und für die Basenpaarung mit dem zweiten Ende zur Verfügung stehen. Durch solch einen Mechanismus wären keine Crossoverprodukte möglich.
2001 wurden die beiden konkurrierenden Modelle DSBR und SDSA dann gleichzeitig von zwei Gruppen zusammengefasst. In dem heute als revised model bezeichneten Prozess nach in der Bäckerhefe gewonnenen Ergebnissen von Hunter und Kleckner (2001) und Allers und Lichten (2001) beginnt die homologe Rekombination zunächst, wie ich es schon für DSBR und SDSA beschrieben habe. Die Aufteilung in die beiden Arme CO und NCO erfolgt jedoch schon vor dem dHJ-Intermediat, nämlich auf Ebene des D-Loop. Von hier ab kann dann die Rekombination entweder zum NCO aufgelöst werden, per SDSA-Modell. Oder eben über die doppelte Holliday Junction zum CO, das nach diesem Modell das einzige Ergebnis des DSBR-Weges ist.

revised model der homologen Rekombination nach Hunter und Kleckner (2001) und Allers und Lichten (2001). Klicken für größere Version.
Auf diesem Stand möchte ich es dann auch belassen für heute. Worauf ich hier überhaupt nicht eingegangen bin, sind die vielen Proteine, die diese ganzen Wege bevölkern. Von manchen kennt man recht gut ihre Position im Schema und ihre dortige Aufgabe, von vielen anderen weiß man aber nur ungefähr, in welche Hälfte des Modells sie passen.
Im nächsten richtigen Post dieser kurzen Serie werde ich dann, bewaffnet mit dem Hintergrund zur homologen Rekombination hier, Untersuchungen unserer Mutanten zur Rekombinationsrate vorstellen.
[1] Vor der Zeit der schnellen Sequenzierung, und auch vor dem Triumphzug der PCR waren solche Marker oft Sporenfarbgene der untersuchten Pilze, oder Antibiotika-Resistenzgene.
JW Szostak, TL Orr-Weaver, RJ Rothstein, FW Stahl (1983). The double-strand-break repair model for recombination Cell, 33 (1), 25-35 DOI: 10.1016/0092-8674(83)90331-8
N Nassif, J Penney, S Pal, WR Engels, GB Gloor (1994). Efficient copying of nonhomologous sequences from ectopic sites via P-element-induced gap repair. Mol Cell Biol., 14 (3), 1613-1625
Neil Hunter, Nancy Kleckner (2001). The Single-End InvasionAn Asymmetric Intermediate at the Double-Strand Break to Double-Holliday Junction Transition of Meiotic Recombination Cell, 106 (1), 59-70 DOI: 10.1016/S0092-8674(01)00430-5
T Allers, M Lichten (2001). Intermediates of Yeast Meiotic Recombination Contain Heteroduplex DNA Molecular Cell, 8 (1), 225-231 DOI: 10.1016/S1097-2765(01)00280-5
Sonntag, März 01, 2009
Gutes für die Ohren 2
Über den Radio Lab Podcast hab ich schon ein paar Mal berichtet. In der aktuellen Folge geht es - um wen sonst? - um Darwin. Wer hören will, wie Charles Darwin und Francis Crick (ja, der mit der Struktur der DNA) miteinander verbunden sind, muss sich aber die halbe Stunde komplett anhören. Ich verrate nicht wann diese nette Info kommt!
WNYC' Radio Lab: Darwinvaganza [MP3-Link, 27:25]
Nach so viel Wissenschaft will man aber auch gut unterhalten werden. Dafür kann ich die letzten drei Folgen StarShipSofa empfehlen, in denen die nominierten Kurzgeschichten der diejährigen British Science Fiction Awards zu hören sind. Besonders Ted Chiang halte ich für einen der besten Autoren, die wir zur Zeit haben. In "Exhalation" schafft er es, Roboter, Anatomie und freien Willen mit Anspielungen auf den "Big Freeze" und der Multiversum-Hypothese zu verbinden. Großartig!
Und wenn diese Kurzgeschichte für mich jetzt schon als Gewinner feststeht, sind die anderen beiden Nominierten auch empfehlenswert. Da wäre einerseits M. Rickerts "Evidence of Love in a Case of Abandonment" über eine Welt, in der die Pro-Life-Fraktion gewonnen hat, und andererseits Paul McAuleys "Little Lost Robot" über einen enormen Roboter, der unsere Galaxis durchfliegt und alles Leben plattmacht.
StarShipSofa
Ted Chiang: "Exhalation" [MP3-Link, 46:59]
M. Rickert: "Evidence of Love in a Case of Abandonment" [MP3-Link, 30:41]
Paul McAuley: "Little Lost Robot" [MP3-Link, 40:23]
Montag, Februar 23, 2009
neues Blog: Labtutorials in Biology
Spannend ist beispielsweise der Post "Liquid handling with pipettes", in dem er mit vielen (auch eigenen) Bildern und Videos das Funktionsprinzip von Pipetten erklärt, die große Vielfalt der verschiedenen Typen von Pipetten - von der Einmalpipette aus Plastik bis zum Pipettierroboter - zeigt, und auf ihren Einsatz im Labor eingeht.
Den Post sollten sich vielleicht mal die vielen Kriminallabor-Techniker aus dem Fernsehen gründlich antun, dass ich nicht mehr diese chronischen Wunden auf dem Unterarm habe [1].
[via ScienceRoll]
[1] Vom ständigen Reinbeißen, um nicht laut aufschreien zu müssen angesichts dem regelmäßigen Schrotten von teuren Pipetten.
Sonntag, Februar 22, 2009
Diamonds in the Sky
Sehr oft ist die Menge echter "science" in Science Fiction eher gering. Da wird überlichtschnell gereist, wenns sein muss auch in die Zukunft oder Vergangenheit. Große Raumschlachten mit Explosionen und Schreien. Die Wissenschaft bleibt zugunsten der Story auf der Strecke. Und während manche Leute sogar der Meinung sind, gute Science Fiction braucht schlechte Science, hat Mike Brotherton für seine Anthologie "Diamonds in the Sky" genau das Gegenteil versucht: SF-Kurzgeschichten zusammenzustellen, die astronomische Zusammenhänge möglichst korrekt wiedergeben. Und zwar so korrekt, dass sein Projekt von der National Science Foundation gefördert wurde, und die Kurzgeschichten für Schüler und Studenten als Lernhilfe dienen sollen!
Sehr schön finde ich, dass in der Anthologie beide Seiten zu Wort kommen - ausgezeichnete Science Fiction-Autoren wie Jeffrey Carver, David Levine oder Mary Robinette Kowal, aber eben auch Wissenschaftler wie Kevin R. Grazier (beteiligt an der Cassini/Huygens Mission), oder Valentin Ivanov (Mitarbeiter des ESO).
Alle Kurzgeschichten sind übrigens frei zugänglich über die Read-Links unten. Viel Spaß beim Lesen!
Contents
In the Autumn of Empire (Jerry Oltion)
A cautionary tale about why scientific misconceptions can be important. This story will also be appearing in Analog soon. Keywords: The seasons. Misconceptions.
ReadEnd of the World (Alma Alexander)
Nothing is forever, not even the earth and sky. Keywords: Evolution of the sun.
ReadThe Freshmen Hookup (Wil McCarthy)
An exploration of how the elements are built in stars using the antics of college freshmen as a metaphor. Keywords: Stellar nucleosynthesis.
ReadGalactic Stress (David Levine)
You think your life is stressful? How about having to deal with the entire universe? Keywords: Scales of the Universe.
ReadThe Moon is a Harsh Pig (Jerry Weinberg)
Robert Heinlein’s novel The Moon is a Harsh Mistress about a revolt on the Moon was a landmark novel of the 1960s. Jerry’s story is also educational. Keywords: Phases of the Moon, Misconceptions.
ReadThe Point (Mike Brotherton)
What is the meaning of life in an expanding universe? This story previously appeared at www.mikebrotherton.com. Keywords: Cosmology
ReadSquish (Dan Hoyt)
How would you like a whirlwind tour of the planets? Keywords: The Solar System.
ReadJaiden’s Weaver (Mary Robinette Kowal)
So many things about life on Earth depend on the cycles of the sky, from the moon and tides to seasons and more. Well, what if the sky were different? How would humans adapt to life on a world with rings? Keywords: Planetary rings
ReadHow I Saved the World (Valentin Ivanov)
The movies Armageddon and Deep Impact featured nuclear bombs to divert asteroids headed for Earth, but this is really not the best way to deal with this threat. This story was originally published in Bulgaria, in the annual almanac “Fantastika”, the 2007 issue. Publisher: “Human Library Foundation”, Sofia. ISSN 1313-3632. Editors: Atanas P. Slavov and Kalin Nenov. Keywords: Killer asteroids
ReadDog Star (Jeffrey A. Carver)
It permeates space and has a subtle but important effect on our existence. What if the effect were not so subtle? Keywords: Dark Energy
ReadThe Touch (G. David Nordley)
Life in the Milky Way can be harsh depending the neighborhood you live in. You should hope you have helpful neighbors when the times are harsh. This story originally appeared in The Age of Reason, edited by Kurt Roth, at SFF.net in 1999. Keywords: Supernova (type 1a)
ReadPlanet Killer (Kevin Grazier and Ges Seger)
And sometimes the times are harsh but you have to depend on yourselves. It helps if you have a little unlikely but useful faster-than-light starships as in Star Trek. Keywords: That would be telling!
ReadThe Listening-Glass (Alexis Glynn Latner)
What’s the future hold for astronomy and astronomers? What would it be like to work on the moon? An earlier version of the story was first published in the February, 1991 issue of Analog Science Fiction/Science Fact. Keywords: Radio astronomy, the Moon
ReadApproaching Perimelasma (Geoffrey A. Landis)
A sophisticated tale about the ultimate journey. Previously published in Asimov’s Science Fiction, Jan. 1998. Keywords: Black holes
Read
Freitag, Februar 20, 2009
Epigenetik ≠ Lamarckismus
Es geht in dem Artikel um aktuelle Forschungsergebnisse aus der Neurobiologie von Mäusen. Kurz zusammengefasst wurde eine Mäuselinie, die aufgrund einer Mutation Schwierigkeiten mit Erinnerungen hat, auf Gedächtnisleistungen untersucht. Die Forscher stellten dabei fest, dass junge Mäuse dieser Linie, wenn sie in einer Umwelt groß gezogen wurden, die das Hirn fordert - Spielzeuge, soziale Interaktionen, Bewegungsfreiheit - besser in Gedächtnistests abschnitten. Soweit so altbekannt. Überraschend kam nun für die Forscher, dass auch die Nachkommen dieser besonders geförderten Mäuse bessere Erinnerungen hatten, auch wenn sie selbst nicht in einer stimulierenden Umwelt aufwuchsen.
Der Autor des Heise-Artikels sieht darin die verspätete Rache von Lamarck an Darwin. Wieso ist das aber falsch, und wie sollten die Ergebnisse eher interpretiert werden?
Jean-Baptiste de Lamarck (1744 - 1829) war ein französischer Naturforscher mit einem recht interessanten Leben. Ohne ein abgeschlossenes Studium verfasste er das damalige Standardwerk über die Flora Frankreichs, Flore françoise, und wurde daraufhin Mitglied der Pariser Akademie der Wissenschaften und Mitarbeiter des Pariser Botanischen Gartens. Die Stelle am botanischen Garten war aber sehr schlecht bezahlt (je nach Quelle sogar gar nicht), er musste also anderweitig für ein Einkommen sorgen: mit dem Veröffentlichen weiterer botanischer Bücher. Dies spielte sich während der französischen Revolution ab, und während anderswo in der Stadt Köpfe abgeschlagen wurden, erhielt Lamarck eine Anstellung und Professur am neugegründeten naturhistorischen Museum - zuständig nun für Insekten und Würmer, aber bevor man sich in dieser Zeit gegen so eine Anweisung von oben wehrt, lernt ein Botaniker doch lieber etwas neues über Tiere.
Bekannt ist Lamarck jedoch bis heute für seine um 1800 entwickelte Evolutionstheorie. Denn während der Gedanke der Evolution, also der Bildung neuer Arten aus älteren, schon längere Zeit in der frühen Naturwissenschaft herumgeisterte [1], fehlte immer noch ein brauchbarer Mechanismus, der dies bewerkstelligen sollte. Nach Ansicht Lamarcks war dies ein allen Organismen innewohnender Drang, sich von einer einfachen Urform zu einer besseren, komplexeren Form hin zu entwickeln.
Das bekannteste Bild, das die Lamarcksche Evolution beschreibt, nimmt die Giraffe zum Beispiel. Warum haben Giraffen so lange Hälse? Weil zunächst Giraffen ihre noch kurzen Hälse ihr ganzes Leben über langgestreckt haben, um die saftigen Blätter ganz oben in den Bäumen zu erreichen, und diese so verlängerten Hälse an ihre Nachkommen vererbten. Nach mehreren Generationen hatte man dann Giraffen mit langen Hälsen. Problematisch wurde für den Lamarckismus, dass Darwin 50 Jahre später auch eine Idee für einen evolutionären Mechanismus hatte, und durch die natürliche Selektion ließen sich nicht nur unzählige Daten aus Fossilien und rezenten Arten erklären, sondern auch die langen Hälse der Giraffen: Es hatte mit den saftigen Blättern ganz oben in den Bäumen zu tun, da lag Lamarck noch richtig. In der Population von Giraffen gab es unterschiedliche Halslängen (die Variation), und wenn nur die Giraffen mit den längsten Hälsen die saftigen Blätter erreichten, dann hatten sie auch mehr Nachkommen, die die langen Hälse erbten.
Wenn es in den Medien in den letzten Monaten um Epigenetik geht, dann wird oft der Lamarckismus heraufbeschworen. Was ist Epigenetik?
Eine genaue Definition von Epigenetik steht noch aus, ganz allgemein versteht man darunter aber vererbbare Eigenschaften, die nicht in der Sequenz der DNA kodiert sind (das wäre die Genetik). Eine epigenetische Vererbung wäre es beispielsweise, wenn die Expressionsstärke eines Gens vererbt wird (in den Extremen also, ob ein Gen an- oder ausgeschaltet ist). Wie könnte so eine Information weitergegeben werden, wenn sie nicht in Form einer DNA-Sequenz vererbt wird? Von den verschiedenen bisher gezeigten Möglichkeiten will ich zwei hier kurz beschreiben: Ein Gen besteht nicht nur aus der Sequenz, die später in ein Protein übersetzt wird. Vor diesem Bereich liegt ein Abschnitt DNA, der sozusagen als (Dimm-)Schalter die Menge an hergestelltem Protein einstellt, indem regulatorische Proteine daran binden - der Promotor. Durch Anfügen von Methylgruppen an Cytosine (eine der vier Basen der DNA) können Promotoren stillgelegt werden. Dies ist keine Änderung der DNA-Sequenz, die Cytosine sitzen immer noch an ihrem Platz im Promotor. Die Methylgruppe ist jedoch kovalent an das Cytosin gebunden, wird also auf dem üblichen Weg mit dem Cytosin an die Nachkommen vererbt. In den Nachkommen wird dieses Gen darum auch stillgelegt sein.
Beim zweiten epigenetischen Mechanismus ist keine Veränderung von Basen nötig. Im Zellkern schwimmt die DNA nicht nackt herum, es sind vielmehr zahlreiche strukturelle Proteine an sie gebunden. Sehr wichtig sind hier die Histone, die in kleinen kugelförmigen Komplexen (Nukleosomen genannt) mit DNA umwickelt sind [2]. Dieses Aufwickeln der DNA ist nötig, um mehrere Meter davon in einem Zellkern mit wenigen Mikrometern Durchmesser unterzukriegen. Nun gibt es mehrere Kondensationsstufen, die stärkste davon die bekannten Metaphasechromosomen. Die Expression von Genen ist in einem so kondensierten Zustand aber nicht mehr möglich.
Die Regulation dieser Kondensation erfolgt über Modifikation der Histone; Anfügen von Methylgruppen erhöht beispielsweise den Kondensationsgrad, während eine angehängte Acetylgruppe ihn verringert [3]. Es ist für die Zelle dadurch möglich, die Expression von Genen in einem relativ kleinen Bereich eines Chromosoms zu regulieren.
In den letzten Jahren wurden nun zahlreiche Hinweise für eine epigenetische Vererbung von "Eigenschaften" gefunden, die sich ein Organismus während seines Lebens aneignet. Die Gedächtnisleistungen von Mäusen im oben verlinkten Heise Artikel sind ein Beispiel dafür, aber auch erhöhte Rekombinationsraten von Arabidopsis-Pflanzen nach UV-Bestrahlung ihrer Eltern fallen darunter. Ganz oberflächlich kann man verstehen, warum manche Leute bei der Vererbung von angeeigneten Merkmalen an Lamarck denken, oder warum bei der Diskussion von Epigenetik in den Medien oft Lamarckismus eingeworfen wird. Es gibt aber mehrere Gründe, warum man diese beiden Begriffe nicht gleichsetzen kann!
- Während bei allen aktuell beschriebenen Forschungen ein äußerer Einfluss das vererbte Merkmal in den Organismen erzeugte, wehrte sich Lamarck vehement gegen solche Umwelteinflüsse auf die Vererbung. Seiner Ansicht nach wohnte schließlich jedem Organismus eine Triebkraft inne, die ihn zum komplexer evolvieren zwang.
- Nach Lamarck akkumulieren sich solche erworbenen Merkmale in einer Population, weil sie nicht verloren gehen können. Epigenetik ist aber ein sehr dynamischer und instabiler Prozess, Effekte wie die im Heise Artikel beschriebenen gehen nach mehreren Generationen verloren.
- Epigenetik ruht immer noch auf einer genetischen Basis. Die epigenetischen Modifikationen werden von Proteinen erzeugt, die ganz herkömmlich im Genom kodiert sind. Epigenetisch vererbte Merkmale unterliegen auch den evolutionären Kräften wie Selektion und Genetic Drift. Also Darwin WIN, Lamarck FAIL.
Witziger Punkt nebenbei: In Technology Review wird auch über eine zweite, ähnliche Studie bei Mäusen berichtet, bei der schlechte Mütter Nachkommen hatten, die auch schlechte Eltern waren. Davon bleibt im Heise Artikel nur noch die Möglichkeit, "dass beispielsweise die Auswirkungen einer frühen Kindesmisshandlung die Generationen überspringen können [...]."
Achja, und beide Daumen hoch für die vielen Kommentatoren zu dem Artikel, die nicht nur die Qualität des Artikels kritisieren, sondern auch die unvermeidlichen Kreationisten in ihre Schranken weisen.
[1] Beispielsweise hat Darwins Großvater Erasmus Darwin darüber ein schönes Gedicht verfasst.
[2] Histonproteine sind positiv geladen, DNA negativ.
[3] Ganz so einfach ist das nicht. Der Effekt ist unter anderem abhängig von dem veränderten Histon, der Aminosäure, die verändert wird, und eben der Art der angefügten Gruppe. In Anlehnung an den genetischen Code wurde diese Regulation als Histon-Code bezeichnet.
Donnerstag, Februar 19, 2009
Neues Spielzeug: Starmap
Daraufhin hab ich mein bisher teuerstes Programm für den iPod touch gekauft: Starmap. Die 10 Euro waren aber gut angelegt, Starmap ist davon jeden Cent wert (und mehr)! Ich will hier nicht alle Funktionen auflisten, dazu habe ich das Programm bisher auch zu wenig genutzt, um alle zu kennen. Deshalb ein eher subjektiver Eindruck, was mir bei der Benutzung gefallen hat.
Starmap braucht die eigene Position, um ein korrektes Bild des Nachthimmels am aktuellen Standort darstellen zu können. Mit einem iPhone wäre das kein Problem, das hat schließlich GPS. Ich musste aber nicht schnell die Längen- und Breitengrade meines Standortes raussuchen, denn auch der iPod touch kann manchmal seine Position finden - über WLAN Hotspots. Und da mein eigener WLAN Router in irgendeiner Datenbank hinterlegt ist (weiß eigentlich jemand, wie Hotspots da rein kommen, und wer die Datenbank betreibt?), fand Starmap alleine die aktuelle Position. Über die aktuelle Uhrzeit wusste Starmap dann auch, welche Objekte gerade am Himmel sein mussten [1].
Was mache ich jetzt, wenn ich beispielsweise das Sternbild Orion finden möchte [2]? Ein Druck auf "Sternbilder" in der Menüleiste bringt mich in eine Liste, die ich zwischen allen und den aktuell sichtbaren Sternbildern umschalten kann. Wenn ich jetzt auf "Orion" drücke, dann erscheint auf der Himmelskarte ein Pfeil, der mich in Richtung Orion lotst, ausgehend von einer Blickrichtung nach Norden.
Die Bewegung dahin registriert der iPod übrigens. Durch die eingebauten Accelerometer kann ich den iPod horizontal und vertikal bewegen, und die Himmelskarte scrollt in die entsprechende Richtung! Rein- und rauszoomen geht übrigens auch wie für den iPod touch/iPhone üblich.
Andere nette Ideen, die sehr clever gelöst sind: Man kann stufenlos sowohl die Helligkeit des Himmels, als auch die der Sterne einstellen, um die Anzeige am Bildschirm den tatsächlichen Sichtverhältnissen anzupassen. Um sich selbst nicht den Blick mit einem hellen Bildschirm zu verschmutzen, gibt es auch einen Rotlichtmodus. Für viele Objekte sind zusätzliche Informationen hinterlegt, für die Planeten auch Bilder. Oh, und Pluto ist zumindest in Starmap noch ein Planet. ;-)
[1] Auch solche Daten wie Auf- und Untergangszeiten sind offline verfügbar, Starmap benötigt keine Internetverbindung.
[2] Entsprechend kann man auch bei Planeten, Sternen und Galaxien vorgehen.
Montag, Februar 16, 2009
Fringe sperrt die Wissenschaftler weg
In der aktuellen Folge 14 scheint man gleich zu Beginn der Frage nachzugehen, wohin man die vielen kriminellen Wissenschaftler wegsperren soll. Klar doch, wir machen am besten ein Gefängnis auf, in das die Abertausende von Bond-Schurken geliefert werden. Nur wo stellt man so ein Gefängnis hin? Das ist eigentlich auch keine schwere Frage, nach Deutschland natürlich! Und der Name "Wissenschaft Prison" kommt beim amerikanischen Publikum sicher klasse an - das klingt so schaurig schön nach Karloff, oder gleich Mengele.
Sonntag, Februar 15, 2009
Alles Gute nachträglich, Charles!
Das Max-Planck-Institut für evolutionäre Anthropologie in Leipzig hat in einer Pressemeldung (Streaming-Video) die Entwurfsequenz des Neanderthalergenoms bekannt gegeben. Das ist erstmal ein wenig bedenklich, denn das Ankündigen von Ergebnissen in der Presse vor einer vollwertigen Publikation in einem peer review-Journal ist kein vorbildliches Handeln für einen Wissenschaftler. Hier kann man aber zumindest ein Auge zudrücken, denke ich - der Termin an Darwins Geburtstag war einfach zu verlockend, außerdem erfolgte die Pressekonferenz simultan auch auf dem Jahrestreffen der American Association for the Advancement of Science (AAAS), die unter anderm das Science Magazine herausgibt. Trotzdem, ein komisches Gefühl in der Magengegend bleibt.
Davon mal abgesehen ist das Ganze natürlich sehr spannend. Erst vor drei Jahren kündigte Svante Pääbo von der Abteilung Evolutionäre Genetik des MPIs an, das Neanderthalergenom sequenzieren zu wollen. Dies ist nur deshalb in so kurzer Zeit möglich (zur Erinnerung: die Arbeiten für das Humangenomprojekt in den 90ern dauerten rund 10 Jahre), weil mittlerweile neue, schnellere Sequenziermethoden zur Verfügung stehen. Diese sog. Sequenziertechnologien der zweiten Generation leiden aber alle an dem Problem, dass sie nur sehr kurze Sequenzstücke (20-50 Basenpaare) produzieren. Und hier hilft dann die harte Vorarbeit des Humangenomprojekts, denn mit dem menschlichen Referenzgenom lässt sich das Neanderthalergenom auch mit sehr kurzen Sequenzen zusammensetzen. Man erwartet nur sehr wenige Unterschiede zwischen den zwei Genomsequenzen, die Genome von Mensch und Schimpanse sind schließlich schon sehr ähnlich.
Um an Neanderthalersequenzen zu kommen, müssen jedoch erstmal mehrere Probleme vermieden werden. Verunreinigungen der Proben mit menschlicher DNA der beteiligten Forscher wären fatal, deshalb mussten die Neanderthaler-Proben in Reinraumbedingungen bearbeitet werden. Trotzdem lagen die Knochen sehr, sehr lange im Boden rum, die Proben enthalten darum jede Menge mikrobiolle DNA. Die wurde natürlich zunächst mitsequenziert, konnte aber dank bioinformatischer Methoden wieder entfernt werden: Bakterielle Genome sind im Vergleich mit dem menschlichen sehr klein, und es wurden bereits viele sequenziert. Indem die Leipziger Forscher ihre Sequenzen mit bakteriellen Genomsequenzen verglichen, konnten sie Verunreinigungen in ihren Daten erkennen und diese dann verwerfen. Diese Verunreinigungen machen ca. 90% des sequenzierten Materials aus! Wenn man dann noch bedenkt, dass die Gruppe um Svante Pääbo bei der DNA-Isolierung wegen fehlerhafter Protokolle ca. 99% Verlust hatte, dann ist es schon erstaunlich, wie weit sie in der kurzen Zeit gekommen sind.
Es muss aber ausdrücklich betont werden, dass es sich bestenfalls um eine erste Entwurfsequenz handelt. Ungefähr 60% des Genoms sind sequenziert, das aber nur einmal. Um sich gegen methodische Fehler der Sequenzierungstechnologien zu schützen, sollte jedes Nukleotid in einer Genomsequenz aber mehrmal sequenziert werden (die menschliche Referenzsequenz hat übrigens 12x Abdeckung). Ein weiteres Problem sind Sequenzunterschiede zwischen Individuen einer Art. Um die Variabilität der Genomsequenz einer Art einschätzen zu können, sollten möglichst viele individuelle Sequenzen vorhanden sein. Dies ist beim menschlichen Genom gerade im Gange, etwa mit dem Personal Genome Project oder dem 1000 Genomes Project. Sogar das erste menschliche Referenzgenom stellt in Wirklichkeit eine Mischung aus den Sequenzen von zwölf anonymen Individuen dar. Demnächst soll aber auch die Sequenzierung weiterer Neanderthaler-Proben beginnen, so dass dieses Problem wohl irgendwann behoben sein sollte.
Ein großes Problem soll auch nicht ungenannt bleiben: Durch das große Alter der Proben ist die DNA stark fragmentiert; es ist darum sehr wahrscheinlich, dass wir nie die komplette Genomsequenz des Neanderthalers erhalten werden.
Trotzdem ist es bereits mit diesen ersten Daten möglich, interessanten Fragen über das Verhältnis von Homo sapiens und Homo neanderthalensis nachzugehen. In weiten Teilen Europas lebten beide Menschenarten für mehrere tausend Jahre nebeneinander. Kam es zu einer Hybridisierung, also zu gemeinsamen Nachkommen beider Arten? Hat der heutige Mensch vielleicht sogar Neanderthaler als Vorfahren? Die bisherigen Sequenzdaten (beruhend auf mitochondrialen Sequenzen und dem Y-Chromosom) deuten eher in Richtung Nein.
Die Ausprägung des Gens MC1R (Melanocortinrezeptor 1), das das Ausmaß der Hautpigmentierung beeinflusst, zeigte, dass es möglicherweise auch hellhäutige und rothaarige Neanderthaler gab (Lalueza-Fox C et al (2007), Science 318:1453-5).
Abschließen möchte ich diesen Post mit (ja, richtig geraten) - einem Podcast! Vor ungefähr zwei Jahren, also noch relativ früh im Neanderthalergenomprojekt, wurde Svante Pääbo zusammen mit Thomas Jarvie (von 454 Life Sciences, die die Sequenziertechnologie zur Verfügung stellen) im immer guten Futures in Biotech Podcast interviewt [MP3-Link]. Ich habe das Interview seitdem nicht mehr gehört, werde aber vielleicht noch mal reinhören. Mal sehen, wie Pääbos Pläne von vor zwei Jahren mit der tatsächlichen Entwicklung mithalten konnten.
[1] Die öffentliche Aufmerksamkeit, die Darwin und Evolution gerade genießen ist sicher richtig und gut, aber ich freu mich auf die nächsten, ruhigeren Wochen. Bis es im Spätjahr dann mit dem 150jährigen Jubiläum der Origin-Veröffentlichung wieder rundgeht.
Freitag, Februar 06, 2009
Kaffee kreiert Krebs?
‘Although there’s no evidence at all of a link between caffeine and cancer, we’re putting two and two together and saying: caffeine can induce these changes and it has been shown that these changes are elevated in leukaemia patients,’ added Dr Cooke.Er leitet sich also aus dem Fehlen eines Beweises die Grundlage seiner Forschung und gleich auch noch das passende Ergebnis ab? Ich ruf da mal gleich beim Nobelpreiskomitee an...
Worauf die bloggenden Kollegen (mit Ausnahme von Neuroskeptic) in ihrer Aufregung aber gar nicht eingegangen sind, und Herr Cooke wohl "übersehen" hat: Die Verbindung Koffein und Genomstabilität ist bereits recht gut verstanden! Leider aber nicht so, wie es in dem Artikel dargestellt wird. Aber der Reihe nach.
Signalisierung von Schäden an der DNA
Ich bin in ein paar Posts schon auf die Reparatur von DNA-Schäden eingegangen, aber einen wichtigen Teil davon habe ich bisher unterschlagen. Denn bevor die Zelle wirklich die Schäden repariert, muss sie erst noch entscheiden, ob sich die Reparatur überhaupt lohnt. Denn bei zahlreichen Schäden besteht immer auch die Gefahr, dass sich Mutationen einschleichen. Und bevor sich dann Krebs entwickelt, opfert der Organismus lieber eine Zelle. Dieser programmierte Zelltod folgt einem strengen genetischen Programm und wird als Apoptose bezeichnet. Hat sich die Zelle entschieden, die Schäden doch zu reparieren, dann müssen zunächst die zahlreichen Reparaturproteine aktiviert werden. Außerdem sollte während der Reparatur der Zellzyklus angehalten werden. Es wäre nämlich sehr schlecht, wenn die Zelle mitten im Reparaturprozess anfängt, ihre DNA zu replizieren, oder sich zu teilen.
ATM und ATR
Eine zentrale Rolle bei all diesen Signalen spielen zwei verwandte Proteine, die beim Menschen ATM und ATR heißen. Beide sind Kinasen, also Enzyme, die die Aktivität anderer Proteine regulieren, indem sie Phosphatgruppen auf bestimmte Aminosäuren übertragen. Im Fall von ATM und ATR ist diese Kinaseaktivität nun abhängig von verschiedenen Schäden an der DNA: Dadurch werden sie aktiviert, und übertragen Phosphate auf Proteine, die die oben angesprochenen Prozesse wie Apoptose, Zellzyklus oder Reparatur regulieren. Fallen ATM und ATR aus, führt dies beim Menschen zu Erbkrankheiten wie Ataxia-telangiectasia (ATM, versucht mal das mehrmals hintereinander zu sagen) oder Seckel Syndrom (ATR). Eigentlich sollte es nicht überraschen, dass Menschen, die an Ataxia-telangiectasia leiden, sehr strahlenempfindlich sind. Ionisierende Strahlung erzeugt schließlich DNA-Schäden, und durch den Ausfall von ATM funktioniert dann die Regulation von Reparatur etc. nicht mehr. Dieser Punkt ist aber noch aus einem anderen Grund wichtig, wie wir gleich sehen werden.

Abbildung des ATM-Signalweges in der Zelle aus dem GeneAssistTM Pathway Atlas von Ambion/Applied Biosystems. Gezeigt sind die wichtigsten Prozesse, die von ATM reguliert werden. Zielprotein werden von ATM aktiviert durch Anheften von Phosphatgruppen, dargestellt durch die Kreise mit "P" an den Proteinen.
Koffein hemmt ATM und ATR
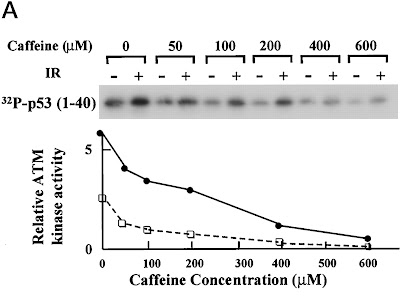
Vor ein paar Jahren machten dann ein paar Forschergruppen eine interessante Entdeckung: Die Aktivität von ATM (und ATR) kann gehemmt werden durch Gabe von Koffein. Die Gruppe von Zhou et al. hat Glück, ihr im Journal of Biological Chemistry veröffentlichter Artikel darf für Lehrzwecke auch online frei benutzt werden. Durch Arbeiten mit Zellkulturen konnten sie zeigen, dass durch die Zugabe von Koffein mehrere von ATM aktivierte Signalketten unterdrückt wurden, beispielsweise ein Stopsignal im Zellzyklus, das die Einleitung der Zellteilung bei DNA-Schäden verhindern soll (der G2/M-Checkpoint). Dieser Effekt ahmt das Verhalten von A-T Zellen (Zellen von Patienten, die an Ataxia-telangiectasia leiden und in denen ATM inaktiv ist) nach, wie in Abbildung 5 des Artikels schön zu sehen ist.
Chk2 ist ebenfalls eine Kinase, die aber hauptsächlich den Reparaturweg reguliert und von ATM aktiviert werden muss. Ohne jetzt groß das Prinzip hinter elektrophoretischen Methoden erklären zu wollen, sieht man nach Bestrahlung der Zellen die Aktivierung von Chk2 durch ATM in der höheren Bande bei 2 (vgl. mit 1), weil das Chk2-Molekül durch Anheften einer Phosphatgrupppe größer geworden ist. Diese Aktivierung findet nach Behandlung mit Koffein nicht mehr statt (die Bande ist bei 4 auf der gleichen Höhe wie die inaktive Kontrolle bei 1, es wurde also keine Phosphatgruppe drangehängt). Dieses Ergebnis ist vergleichbar mit dem Verhalten von A-T Zellen ohne Koffeinbehandlung (Nummern 5/6).
In Abbildung 7 haben Zhou und Kollegen einen weiteren von ATM regulierten Weg angesehen, die Apoptose. Sehr wichtig zum Einleiten der Apoptose (dem programmierten Zelltod) ist das Protein p53, das ebenfalls durch Anheften einer Phosphatgruppe von ATM aktiviert werden kann. Wie man hier sehr schön sehen kann, wird p53 mit steigender Koffeinkonzentration immer weniger aktiviert, und das sogar ohne Bestrahlung (ausgeüllte Kreise: +Bestrahlung, offene Quadrate: -Bestrahlung)! Dies bedeutet, dass auch in wenigen Zellen, deren DNA nicht durch Bestrahlung massiv geschädigt wurde, spontane Schäden auftreten, die in einer Aktivierung von p53 (und wohl dem Auslösen der Apoptose) resultieren. Und Koffein kann das durch Hemmung von ATM unterdrücken.
Die medizinische Anwendung
Weiter oben habe ich bei der Beschreibung der Krankheit Ataxia-telangiectasia gesagt, dass die Patienten sehr strahlensensitiv sind, und die Daten hier haben auch gezeigt warum: ATM als zentraler Regulator von vielen zellulären Prozessen nach Schädigung der DNA kann diese Prozesse nicht mehr aktivieren, die Schäden werden nicht repariert und die Zellen versuchen munter, sich trotzdem zu teilen. Da Koffein dieses Erscheinungsbild von A-T Zellen nachahmen kann, wird es seit mehreren Jahren in der Krebsmedizin eingesetzt: Tumorzellen reagieren auf eine Strahlentherapie sensitiver, wenn der Patient zusätzlich Koffein verabreicht bekommt. Und dieser nützliche Effekt konnte auch für mehrere Chemotherapeutika gezeigt werden (etwa Cisplatin).
Nicht vergessen: die Konzentration!
Was in dem Daily Mirror-Artikel leider unterschlagen wird: Um diese Effekte von Koffein beim Menschen zu erreichen, sind Konzentrationen von etwa 10 mM nötig. Nach Trinken einer Tasse starken Kaffees liegt die Konzentration im Blut aber bei etwa 50 µM [1], sie ist also circa 200 mal niedriger. Anders ausgedrückt: Koffein ist nach maximal fünf Stunden im Blut wieder abgebaut, man müsste also mindestens 200 Tassen starken Kaffee innerhalb von weniger als fünf Stunden trinken, um einen Effekt von Koffein auf ATM zu erreichen!
Das Fazit der ganzen Geschichte: Der Daily Mirror sollte sich schämen, schwangeren Frauen Angst einzujagen, und Herr Cooke sollte vor seinem nächsten Projekt zumindest fünf Minuten recherchieren, mehr brauchts nämlich nicht. Und die Literatur ist wie gezeigt zumindest teilweise frei zugänglich.
[1] Berechnet aus einer Koffein-Plasmakonzentration von 10 mg/l und der Annahme von 8 mg/kg Koffein nach Genuss einer Tasse Kaffee.
[2] Sorry für den Titel, aber ich konnte es mir nicht verkneifen ;-)
Zhou, B. B., Chaturvedi, P., Spring, K., Scott, S. P., Johanson, R. A., Mishra, R., Mattern, M. R., Winkler, J. D., Khanna, K. K. (2000). Caffeine abolishes the mammalian G(2)/M DNA damage checkpoint by inhibiting ataxia-telangiectasia-mutated kinase activity J Biol Chem, 275 (14), 10342-10348
Mittwoch, Februar 04, 2009
Die geplatzte Blase
Of course, if you have a mountain of loot you probably want to protect it. Enter the lawyers. Millennium had always filed on their discoveries; now they had lots of discoveries to protect. But protect from what? Well, the paranoia was a loss of "Freedom to Operate", usually known as FTO. Nobody knew what would stand up as a patent -- but there were instructive examples from the early biotech era of business plans sunk by a loss of FTO -- and expensive lawsuits that clearly marked that loss. So the patenting engine took off -- an expensive insurance policy against an unpredictable future.
[...]
This was the late 90's and the hype was getting thick -- we were guilty but so were others. Millennium wasn't a big pusher of high gene counts -- at least in the terms of the day (but that's another whole story), but certainly we started selling all those genes we had & the ones we extrapolated were still out there. A key part of the business model was to sell the genes many times -- if we could sell the same gene to Lilly for cardiovascular & Roche for metabolic and AstraZeneca for inflammation, all the better. Not that anything underhanded went on; we'd present the case to each company & most of the deals had exclusivity only within a therapeutic area.
Auf den Punkt mit der Genzahl ist er dann in einem weiteren Post genauer eingeganen. Woher kamen die unheimlich großen Zahlen in den Schätzungen, wieviele Gene im menschlichen Genom zu finden sind, zu einer Zeit bevor die menschliche Genomsequenz veröffentlicht war? 50 000, 100 000, oder vielleicht sogar 200 000? Wohl hauptsächlich Konkurrenz. Wenn die Biotechfirmen A und B beide mit Bioinformatik und viel Fleiß an den Sequenziergeräten Datenbanken mit menschlichen Genen zusammenstellen und diese verkaufen wollen, etwa an Pharmafirmen, welche Datenbank wird dann eher gekauft? Solange man sich nur auf grobe Schätzungen verlassen kann vorsichtshalber die mit doppelt so viel Genen. So hat sich das dann langsam hochgeschaukelt in sehr luftige Höhen. Selber schuld, wenn alle mitspielen. Umso ernüchternder war für die Kunden dann das Resultat aus dem Human Genome Project: ca. 25 000 bis 30 000 Gene, die in den letzten Jahren dann auf eher 22 000 bis 23 000 zusammengeschrumpft sind.
Das soll aber nicht heißen, dass sich nicht ein paar Leute schon in den Neunzigern darüber Gedanken gemacht hätten:
So my colleague tried a new approach, which I think was to say: we have a few percent of the human genome sequences (albeit mostly around genes of interest and not randomly sampled). How many genes have been found? And what would that extrapolate out to for the whole genome.
His conclusion was so shocking I admit I refused to believe it at first, and never quite bought into it. I think it was about 25-30K. How could the textbooks be off by 2X-3X? I could believe the other genomics companies might be optimistic in interpreting their data, but could they really be deluding themselves that much??
But, the logic was hard to assault. In order for his estimate to be low by a lot, you would have to posit that the genomic regions sequenced to date were unusually gene poor -- and that the rest of the genome was packed.
Beide Artikel sind sehr lesenswert, und schon allein deshalb interessant, weil man über die jungen Jahre der Genomik meistens nur von der Seite der akademischen Forschung hört.
Dienstag, Februar 03, 2009
(leider keine) JoVE Videos
Solche Videos sind aber leider höchstens für Leute interessant, die auf einem ähnlichen Gebiet arbeiten.
So habe ich mir etwa angesehen, wie man Antikörperfärbungen an Hühnerembryonen macht, mit welchem Versuchsaufbau man die Rolle von Geruchssignalen auf den Flug von Drosophilas untersuchen kann, oder auch wie man an methylierte DNA kommt.
Hört sich alles sehr spannend an, ist dann aber nach der kurzen Einleitung schnell recht langweilig. Ich bleibe aufmerksam, glaube aber leider nicht, dass ich so bald wieder über ein spannendes Video berichten kann.
Auslese 2008 - ich bin dabei!

Ich habe während dem Studium aber nur wissenschaftliches Schreiben gelernt, was eine eher trockene, formalisierte Sprache ist. Das Vermitteln von wissenschaftlichen Themen auch und besonders an Nicht-Wissenschaftler etwa hier auf dem Blog stellt mich also vor die Herausforderung, aus meinem üblichen wissenschaftlichen Sprachmuster auszubrechen. Was mir nicht immer gelingt, wie mir selbst bewusst ist.
Umso mehr hat es mich gefreut, dass eine fünfköpfige Jury von Wissenschaftlern und Journalisten meinen Beitrag "Was uns Forschung an Pflanzen über Parasiten sagt" für gut genug hielten, um ihn zusammen mit 14 weiteren sehr empfehlenswerten Blogposts in die Wissenschaftsblog-Auslese 2008 aufzunehmen!
In dem Sinn: Herzlich Willkommen an alle, die vom Wissenschafts-Café hierher gefunden haben. Seht euch ruhig um, und lest auch in andere wissenschaftliche Beiträge von mir rein. Ich werde mich bemühen, auch 2009 Beiträge zu schreiben, die würdig für die nächste Auslese sind.
Vielen Dank an die Juroren, aber auch an Marc und Lars für die Idee und die Organisation - und an den anonymen Nominator, der meinen Beitrag für die Auslese vorgeschlagen hat!
Freitag, Januar 30, 2009
Spielwiese Bioinformatik
Ein sehr gutes Beispiel dafür ist der Genome Projector, der Genomdaten von vielen (bisher nur) Bakterienarten mithilfe dem bekannten Google Maps-Interface darstellt.
Das Referenzgenom des Laborstammes K12 von Escherichia coli (leicht in der Liste zu finden, es ist bereits mit einem Balken markiert) ist ein guter Startpunkt für jeden, der mal einen Blick reinwerfen möchte. Wer kann in der Pathway Map den Citratzyklus finden?
Dienstag, Januar 27, 2009
Und ich sags noch!
Unerwartete EvolutionPressemeldung beim Informationsdienst Wissenschaft
Sonja von Brethorst, Presse- und Öffentlichkeitsarbeit
Stiftung Tierärztliche Hochschule Hannover
26.01.2009
Höhere Tiere stammen nicht von niederen Tieren ab
Wissenschaftler der Stiftung Tierärztliche Hochschule Hannover (TiHo), des Sackler Institute for Comparative Genomics im American Museum of Natural History und der Yale University stellen in der neuesten Ausgabe des Online-Fachmagazins PLoS Biology überraschende Ergebnisse der Evolutionsforschung vor. Die Publikation kann ab Dienstag, den 27. Januar 2009 unter http://biology.plosjournals.org im Internet eingesehen werden.
Die deutsch-amerikanische Arbeitsgruppe hinterfragt mit ihren Forschungsergebnissen die bisherige Auffassung über den Verlauf der Evolution der Tiere. Bislang galt es als selbstverständlich, dass die Evolution der Tiere vom einfachen zum komplexen Tierstamm erfolgte. Die neuen Forschungsarbeiten zeigen jedoch, dass sich die niederen Tiere parallel zu den höheren Tieren entwickelt haben. Zu den niederen Tieren werden beispielsweise Korallen und Quallen gezählt, zu den höheren Tieren gehören alle bekannten Gruppen vom Wurm bis zum Menschen.
Hab ichs nicht gerade erst gesagt? In wenigen Sätzen, sowohl die alte Leier mit der scala naturae, und eine interessante Einteilung der Tiere in zwei Gruppen - höhere und niedere. Hier wird wenigstens die Grenze an einer bekannten Stelle gezogen, mit den höheren Tieren sind die Bilateria gemeint. Aber wieso dann nicht Bilateria sagen?
In den Kommentaren zu meinem letzten Post hierüber habe ich aus Papers zitiert, die die Grenze zwischen höheren und niederen Tieren ganz woanders ziehen, beispielsweise bei den Wirbeltieren. Sinn macht das Ganze jedenfalls keinen.
Und jetzt klaue ich zum Abschluss noch bei T. Ryan Gregory von Genomicron, der sich über das Paper auch aufgeregt hat:
"It is absurd to talk of one animal being higher than another" (Charles Darwin, 1837)

Montag, Januar 26, 2009
Wofür Eliteunis jetzt alles Geld ausgeben können
Ich gehe durchschnittlich einmal pro Tag vom Büro/Labor (der Turm im Hintergrund) zu unserem Versuchsgewächshaus (ein paar hundert Meter hinter mir) und zurück. Bisher konnte man auf dieser Strecke wertvolle Sekunden sparen, indem man zwischen den Bäumen abkürzte. Was auch viele Leute an der Uni machen, der Trampelpfad ist nicht nur von mir! Jetzt hat die Uni wohl was dagegen, dass
Projekt Paperübersicht #2: Der RTR-Komplex in Pflanzen?
Den Großteil unserer Arbeit machen wir mit Insertionsmutanten. Das sind Pflanzen, bei denen mit Hilfe des Bodenbakteriums Agrobacterium tumefaciens relativ große DNA-Fragmente in die Sequenz eines Gens eingeführt wurden. Dies führt im Endeffekt dazu, dass das Proteinprodukt dieses Gens nicht mehr hergestellt werden kann, die Pflanzenlinie unterscheidet sich also von Wildtyppflanzen nur darin, dass ein bestimmtes Gen ausgeschaltet wurde. Der Sinn hinter der Untersuchung von diesen Knockouts ist schnell einleuchtend: Vergleicht man Wildtyp und Knockoutlinie, dann ist sehr wahrscheinlich, dass jeder zusätzliche Defekt in der Knockoutlinie auf dem Ausfall des Gens beruht. Jetzt muss man nur noch umdenken - der Ausfall eines Gens führt zu bestimmtem Defekt, dann erfüllt dieses Gen normalerweise diese bestimmte Funktion, die ein Entstehen des Defekts verhindert.
Zunächst muss man sich natürlich fragen: Gibt es die Gene des RTR-Komplexes überhaupt in Arabidopsis? Hier kann man sich heutzutage sehr viel Arbeit sparen, dank sehr guter Bioinformatik und den frei zugänglichen Genomdatenbanken vieler Organismen. Was wir fanden, passt sehr gut den bereits beschriebenen Genen, vor allem aus der Bäckerhefe Saccharomyces cerevisiae und dem Menschen.
Für das Gen RMI1 (bzw. BLAP75) gibt es im Arabidopsisgenom zwei mögliche Kandidaten. Einer davon (das Gen At5g19950) scheint nicht exprimiert zu werden, und auch von uns untersuchte Insertionsmutanten waren völlig unauffällig. Hierbei könnte es sich also um ein Pseudogen handeln. Der zweite Kandidat (At5g63540) war dafür umso interessanter, wie ich heute und in den folgenden Posts zu unserem Paper zeigen will. Dieses Gen wurde von uns darum AtRMI1 getauft [1].
Auf zum T aus RTR - der Topoisomerase. Hier war es leichter, weil als Homolog zu TOP3 bzw. TOPO3α nur ein mögliches Gen in Frage kam, At5g63920 oder AtTOP3α.
Ein wenig komplizierter wird es bei der RecQ Helikase. Bei dieser Familie wissen wir schon länger, dass es in Arabidopsis sieben Mitglieder gibt. Welche von diesen sieben ist nun aber das funktionelle Homolog zu der einzigen RecQ Helikase in Hefe, SGS1? Im Menschen ist dies unter fünf Familienmitgliedern das Gen BLM, und mit dem Aufbau dieser beiden Gene bewaffnet fallen schon ein paar der Arabdopsis RecQ Helikasen raus. Von den restlichen bleibt dann nur noch RECQ4A (At1g10930) übrig, wenn man bereits bekannte Eigenschaften der Mitglieder der RecQ Familie in Arabidopsis mit einbezieht (Hartung et al., 2007).
Glücklicherweise waren für diese Gene dann auch Insertionsmutanten verfügbar, mit denen wir arbeiten konnten.
Zum Abschluss jetzt noch ein paar "richtige" Daten. Die Beschreibung, wie und warum man bestimmte Gene überhaupt gefunden und benutzt hat, und wie die Mutanten aufgebaut sind, ist zwar ein wichtiger Teil eines Papers - es ist aber eher langweilig.
Es gibt eine relativ einfache Methode, die Beteiligung eines Gens bzw. dessen Proteinprodukts an der DNA Reparatur zu untersuchen: Schädigt man die DNA absichtlich, dann müssen diese Schäden repariert werden. Ist nun aber ein Gen ausgeschaltet, das in die Reparatur der DNA involviert ist, dann funktioniert die Reparatur logischerweise nicht mehr so gut. Das kann man auf verschiedene Weisen messen, wir bestimmen das relative Frischgewicht von behandelten Keimlingen verglichen mit gleich alten unbehandelten Keimlingen. Denn Probleme bei der DNA-Reparatur resultieren beispielsweise im Absterben der betroffenen Zellen, die sich dann auch nicht mehr Teilen oder Wachsen können. Die Folge: Kleinere, leichtere Keimlinge.
Die Bandbreite an möglichen Schäden an der DNA ist unheimlich groß, entsprechend gibt es auch jede Menge von Reparaturwegen. Indem man die Organismen verschiedenen Stoffen mit bekannten Schadeffekten an der DNA aussetzt, kann man mit diesem Versuch auch eine erste Einordnung der untersuchten Gene in diese Reparaturwege vornehmen. Eine Knockoutmutante wird nämlich nur dann sensitiver als der Wildtyp auf einen Stoff reagieren, wenn es in dem betroffenen Reparaturweg aktiv ist.
In dieser Abbildung sind Sensitivitäten von Knockoutmutanten der Gene RECQ4A, TOP3α und RMI1 gegenüber verschiedenen Mutagenen gezeigt. Gegen Methylmethansulfonat (MMS, A in der Abbildung) sind viele Gene der homologen Rekombination sensitiv, so auch unsere drei Gene. MMS erzeugt Addukte an der DNA, indem es eine Methylgruppe an Stickstoffatome in den Basen der DNA hängt. Dies hat zu Folge, dass die normale Doppelhelix verzerrt wird, und elementare Vorgänge wie die Transkription und Replikation nicht mehr stattfinden können. Dies ist wahrscheinlich auch die Verbindung mit der homologen Rekombination, da sie mithilft Probleme direkt an der Replikationsgabel zu beheben. Davon abgesehen können Schäden durch eine Alkylierung aber auch durch die Reparaturwege der Basenexzisionsreparatur und der Nukleotidexzisionsreparatur behoben werden [2]. Die Ungenauigkeit dieser beiden Wege führt übrigens über den Umweg von Mutationen zu einer krebsfördernden Wirkung von MMS.
Cisplatin (genauer cis-Diamindichlorplatin, B in der Abbildung) ist ein interessanter Stoff. Über die eher zufällige Entdeckung dieses kleinen Moleküls habe ich schon mal geschrieben. Heute ist es einerseits eines der am häufigsten eingesetzten Chemotherapeutika, andererseits aber auch eines der bestuntersuchten Stoffe, was die Mechanismen der DNA-Schädigung angeht. Was passiert, wenn Cisplatin in die Zelle kommt? Das zentrale Platinatom reagiert freudig mit so ziemlich jedem Molekül, das es in die Finger kriegt. Deshalb findet man dann Platinaddukte an Zuckern, Fetten, Proteinen, aber eben auch der DNA. Blöd an der Sache ist aber, dass Cisplatin über seine beiden Chloridgruppen auch zweimal reagieren kann, und damit zwei vorher unverbundene Moleküle kovalent verknüpft. So werden dann schon mal Proteine an die DNA gebunden, oder noch schlimmer, zwei Basen der DNA kovalent verbunden. Dies nennt man Crosslinking, und davon gibt es zwei Möglichkeiten: entweder werden zwei Basen aus unterschiedlichen DNA-Strängen verknüpft (=Interstrang-Crosslink), oder es werden zwei Basen innerhalb eines Stranges verknüpft (=Intrastrang-Crosslink). Letzteres ist bei Cisplatin das häufigere Produkt, und auch hier liegen die Reparaturaufgaben bei der Nukleotidexzisionsreparatur und der homologen Rekombination (die besonders während der Replikation).
Zuletzt noch Camptothecin (CPT, C in der Abbildung). Das ist ein Alkaloid, das beispielsweise von der Pflanze Camptotheca acuminata zur Schädlingsabwehr gebildet wird. Und die Schädlinge, die an der Pflanze knabbern tun mir wirklich leid: Im vorigen Teil habe ich knapp beschrieben, was eine Topoisomerase mit der DNA macht. Camptothecin behindert diesen Vorgang der Topoisomerase 1. Genau in dem Zustand, wenn TOP1 kovalent mit der DNA verbunden ist und ein Einzelstrangbruch in der DNA vorliegt, bindet CPT daran und verhindert, dass die Topoisomerase die Lücke schließt und sich von der DNA löst. Dies hat zur Folge, dass bei der Replikation einerseits ein Protein kovalent verbunden auf der DNA sitzt und die Replikationsproteine behindert, und dass außerdem durch das Entwinden des Doppelstrangs aus dieser Lücke in der DNA plötzlich ein Doppelstrangbruch wird. Großes Problem, und sehr wahrscheinlich der Tod des hungrigen Schädlings [3].
Wie man sehen kann, sind alle Mutanten sensitiver als der Wildtyp (Col-0 in der Abbildung) gegenüber MMS und Cisplatin. Gegen CPT ist dagegen nur die Knockoutmutante von TOP3α sensitiv, die beiden anderen nicht. top3A-2 reagiert genau genommen auch auf MMS und Cisplatin sensitiver als recq4A-4 und rmi1-2. Während nun diese gemeinsamen Sensitivitäten eines der Indizien sind, dass die drei Proteine ihre Aufgaben gemeinsam erfüllen (also möglicherweise in einem RTR-Komplex), deuten die Resultate auch auf eine Sonderrolle für TOP3α hin. Auf die will ich aber erst im nächsten oder übernächsten Post eingehen - mit nem Cliffhanger müsst ihr einfach wiederkommen, oder?
[1] So wirklich neue und kreative Namen gibts bei Genen eher selten, aber es kommt vor. Siehe etwa das Gen SUPERMAN und sein Antagonist KRYPTONITE...
[2] Wenn ich jetzt noch anfange, jeden Reparaturweg ausführlich zu beschreiben werde ich gar nicht mehr fertig mit dem Post. Ich hab da aber eine nette Idee: Ich könnte den Mutagenen eigene Posts widmen, und dabei dann auch auf die relevanten Reparaturwege eingehen.
[3] Die Camptothecin-produzierenden Pflanzen besitzen übrigens TOP1-Gene mit leicht veränderten Sequenzen, so dass sie selbst nicht anfällig gegenüber CPT sind (Sirikantaramas et al, 2008).
Montag, Januar 19, 2009
Edgar Allan Poe
It was many and many a year ago,Annabel Lee, von Edgar Allan Poe
In a kingdom by the sea,
That a maiden there lived whom you may know
By the name of Annabel Lee;
And this maiden she lived with no other thought
Than to love and be loved by me.
I was a child and she was a child,
In this kingdom by the sea:
But we loved with a love that was more than love —
I and my Annabel Lee;
With a love that the winged seraphs of heaven
Coveted her and me.
And this was the reason that, long ago,
In this kingdom by the sea,
A wind blew out of a cloud, chilling
My beautiful Annabel Lee;
So that her highborn kinsmen came
And bore her away from me,
To shut her up in a sepulchre
In this kingdom by the sea.
The angels, not half so happy in heaven,
Went envying her and me —
Yes! — that was the reason (as all men know,
In this kingdom by the sea)
That the wind came out of the cloud by night,
Chilling and killing my Annabel Lee.
But our love it was stronger by far than the love
Of those who were older than we —
Of many far wiser than we —
And neither the angels in heaven above,
Nor the demons down under the sea,
Can ever dissever my soul from the soul
Of the beautiful Annabel Lee:
For the moon never beams, without bringing me dreams
Of the beautiful Annabel Lee;
And the stars never rise, but I feel the bright eyes
Of the beautiful Annabel Lee;
And so, all the night-tide, I lie down by the side
Of my darling — my darling — my life and my bride,
In her sepulchre there by the sea,
In her tomb by the sounding sea.
Dies ist das letzte von Poe verfasste Gedicht vor seinem frühen und mysteriösen Tod 1849. Es ist einer der Texte von Poe, die mich am meisten berühren (neben The Raven, aber das Gedicht dürften viele ja kennen). Höchstwahrscheinlich ist es an seine Frau Virginia gerichtet - sie war die letzte in einer ganzen Reihe von Poe nahestehenden Frauen, die er durch Tuberkulose verlor. Eine von Poes Kurzgeschichten, die Maske des Roten Todes (The Masque of the Red Death), bezieht sich wahrscheinlich auch auf die Tuberkulose und die zur Zeit der Veröffentlichung daran leidenden Virginia. Ich finde es faszinierend, dass zwei der wichtigsten Themen von Poes Texten - Frauen und Tod - auch sein Leben auf eine Weise prägten, dass es eine seiner Kurzgeschichten hätte sein können.
In guter Tradition stoße ich mit einem guten Cognac an und hinterlasse drei rote Rosen.
Poe auf Deutsch (Projekt Gutenberg-DE)
Poe auf Englisch (Project Gutenberg)
Poe zum Anhören (Librivox.org)
Donnerstag, Januar 15, 2009
Wieso man zumindest naturwissenschaftliche Kenntnisse der Mittelstufe haben sollte, wenn man medizinische Pressemitteilungen herausgibt...
Dies ist mal wieder ein gutes Beispiel dafür, warum ich normalerweise verzichte, Pressemeldungen zu lesen. Eine Forschergruppe hat in einem aktuellen Paper in Nature gezeigt, dass eine microRNA (miR-21) zur Erkrankung der Herzmuskelschwäche beitragen kann. Das interessante daran ist, dass sie eine Verbindung zwischen einem der vielen Gene Silencing-Mechanismen und einer menschlichen Erkrankung zeigen konnten. Wohlgemerkt, diese Verbindung ist schon länger bekannt, wie die Autoren selbst schon im Abstract des Artikels schreiben. Soweit ist alles noch in Ordnung.
Aber was macht die Pressestelle des an der Arbeit beteiligten Rudolf-Virchow-Zentrum daraus?
"Genetischer Müll" löst Herzmuskelschwäche ausDie Pressemeldung geht noch weiter, befasst sich mit diesem doch scheinbar so hochinteressanten Thema aber nicht mehr. Vielleicht, weil in der Pressestelle nichts tiefschürfenderes als dieses oberflächliche Blabla bekannt war?Würzburg, 30.11.2008. Winzige Bruchstücke des Erbguts - so genannte "micro-RNAs", die bis vor kurzem als unwichtig galten, könnten jetzt die Prävention, Diagnose und Therapie der Herzmuskelschwäche revolutionieren. Würzburger Forscher fanden eine solche erstmals im Herzen, blockierten sie und konnten damit nicht nur gefährdete Mäuse vor dem Ausbruch der Erkrankung schützen, sondern auch an Herzmuskelschwäche erkrankte Mäuse heilen. Die Ergebnisse beschreiben die Wissenschaftler der Universität Würzburg in dem renommierten Wissenschaftsmagazin Nature.Die Entschlüsselung des menschlichen Genoms brachte eine überraschende Nachricht mit sich: Nur ungefähr 1,5 Prozent der gesamten genetischen Information wird für die Herstellung unserer Proteine benötigt, wobei die Gene über eine Art Kopie, die Boten-RNA, in Proteine umgeschrieben werden. Der Rest der Gene galt lange als bedeutungsloser "genetischer Müll", weil deren RNA ohne nennenswerte Funktion sei. 2006 wurde jedoch für die Entdeckung, dass RNAs, aus denen keine Proteine entstehen, auch wichtige Aufgaben im Körper besitzen den Medizin-Nobelpreis verliehen [sic]. Diese RNA-Stücke regulieren die Bildung der Proteine über Bindung an die Boten-RNA direkt. Geht hier etwas schief, produziert der Körper also beispielsweise zu viel oder zu wenig Proteine, gerät die Körperzelle aus dem Gleichgewicht - Krankheiten entstehen.
Gehen wir das Ganze doch von vorne nach hinten durch. Der Titel ist schon super, "Genetischer Müll" gleich in scare quotes, und irgendwie klingt es so, als ob endlich der ultimative Grund der Herzmuskelschwäche gefunden wäre. Auf den genetischen Müll, also die junk DNA, möchte ich gleich ausführlicher eingehen, jetzt wird erst mal diese Meldung zerpflückt.
Erster Satz - microRNAs (schon wieder scare quotes) sind keineswegs "Bruchstücke des Erbguts", und sie galten auch nicht bis vor kurzem als unwichtig. Vielmehr werden microRNAs genauso wie alle anderen Gene (also auch die, die für Proteine kodieren) zunächst mal transkribiert, d.h. es wird eine Abschrift des Gens in Form einer RNA hergestellt. Bei proteinogenen Genen nennt man diese RNA dann mRNA (messenger RNA). Bei Genen für microRNAs, nunja, microRNA halt. Wären es wirklich Bruchstücke der DNA wäre es einerseits microDNA, und andererseits wäre die Zelle tot (Chromosomen zerbrochen und so). Eine schnelle Suche bei Pubmed ergibt außerdem, dass mindestens seit 2001 über microRNAs geschrieben wird - keineswegs "vor kurzem" also. Nehmen wir nicht nur diese Subfamilie der regulatorischen RNAs, sondern den gesamten Prozess des gene silencing, dann wird die Aussage noch lächerlicher. Über post-transcriptional gene-silencing wird dann auch schon seit 1955 geschrieben (mit bisher fast 19000 veröffentlichten Artikeln), und auf der Informationsseite für den (im Pressetext ja ausdrücklich erwähnten) Nobelpreis (Fire und Mello, 2006) wird direkt Bezug genommen auf Arbeiten in Pflanzen und Pilzen von 1990.
Im zweiten Absatz geht es dann munter so weiter. Da widerspricht sich der Text erstmal heftig. Einerseits sollen nur 1,5% des Genoms Gene sein ("für die Herstellung unserer Proteine benötigt"), andererseits ist das restliche Genom aber wohl voll von Genen ("Der Rest der Gene")!
Dieser Satz ist sowieso voll von Fehlern:
Der Rest der Gene galt lange als bedeutungsloser "genetischer Müll", weil deren RNA ohne nennenswerte Funktion sei.Also, keine Definition was ein Gen ist. Eigentlich sogar das Eingeständnis, seit der 7. Klasse nicht mehr im Bio-Unterricht aufgepasst zu haben. Wörtlich genommen geht der Verfasser wohl davon aus, dass diese 98,5% des Genoms (weil sie ja angeblich Gene sind), auch in RNA umgeschrieben werden. Dies passiert zum allergrößten Teil aber nicht - das Genom ist transkriptionell überwiegend tot. Noch wichtiger, bisher hat noch niemand behauptet, dass eine einmal hergestellte RNA keine Funktion habe [1].
Jetzt aber zur junk DNA. Wann immer in den letzten Jahren jemand einen Effekt gefunden hat, der nicht dem Schema Gen - mRNA - Protein folgt, hat er gleich die böse junk DNA ausgepackt. Demnach sind ja alle anderen Molekularbiologen so beschränkt, dass die alles im Genom, was nicht diesem einfachen Schema entspricht, als genetischen Müll abtun, der völlig unnötig rumliegt und die Suche nach Genen erschwert. Jetzt kommt aber der Underdog, der mit seiner tollen neuen Entdeckung dem engstirnigen wissenschaftlichen Establishment entgegentritt. Klar, dass so eine Darstellung von den Medien gerne aufgegriffen wird.
Mit dieser Sichtweise gibt es nur ein Problem: Sie ist falsch, und das schon fast so lange, wie wir überhaupt wissen dass es Gene gibt und dass die DNA das Erbmaterial in der Zelle ist. Nur wenige Jahre nach der Beschreibung der DNA-Struktur durch Watson und Crick 1953 wurde die Translation in ihren Grundzügen aufgeklärt, also das Herstellen von Proteinen aus der Information der mRNA. Hier war aber schon klar, dass für diesen Prozess RNAs benötigt werden, von denen keine Proteine produziert werden: die tRNAs und die rRNAs. Auch das lernt eigentlich jeder in der Schule. Die Grundannahme, nicht-kodierende RNAs mit einer Funktion in der Zelle wären erst seit ein paar Jahren bekannt, ist also nicht nur falsch - diese RNAs gehören zum Establishment!
Das ist der eine Aspekt. Die andere Seite hat mit der Definition des Begriffs junk DNA zu tun. Zunächst mal ist nicht alles "Müll", von dem wir die Funktion nicht kennen. Mit junk DNA werden Elemente im Genom bezeichnet, die nachweislich keine Funktion haben! Der Nobelpreisträger Sydney Brenner gibt in diesem Video auf iBioSeminars (ab ca. 11:20) ein sehr gutes Beispiel. Die Alu Sequenz mit einer Länge von etwa 300 Basenpaaren kommt im menschlichen Genom in mehreren Millionen Kopien vor; sie macht damit je nach Schätzung zwischen 10 und 30% des gesamten menschlichen Genoms aus! Funktion? Fehlanzeige! Letzten Endes sind Alu-Elemente Parasiten von genetischen Parasiten.
Sogenannte Transposons sind genetische Elemente, die bestimmte Sequenzen an ihren Enden tragen und dazwischen Gene kodieren, deren Proteinprodukte diese Endsequenzen erkennen und dort Schnitte in die DNA setzen. Ein Transposon kann so per cut and paste von einer Stelle im Genom an eine andere wandern. Einen Nutzen für das Wirtsgenom hat das erstmal keinen, Transposons nutzen es nur als Transportvehikel. Alu-Elemente und andere, ähnliche Sequenzen gehen dabei noch einen Schritt weiter: Um selbst Gene zu tragen sind sie zu klein. An ihren Ende besitzen sie aber auch die Sequenzen, die von den Transposonproteinen erkannt werden. Alu-Elemente sind also ehemalige Transposons, die sich den Aufwand mit den Genen gespart haben und ihre noch funktionsfähigen Transposonkollegen ausnutzen. Wiederum aber kein direkter Nutzen für die Wirtszelle.
Larry Moran hat in den letzten Monaten verschiedenste Elemente im menschlichen Genom aufgelistet, die gesichert keine Funktion besitzen, also unter junk DNA fallen. Die Alu Elemente sind auch dabei (zu finden unter SINEs). Insgesamt ist er jetzt schon, ohne alle Arten von funktionslosen Elementen im Genom genannt zu haben, bei über 50% des Umfangs des Genoms - und das ist eine sehr zurückhaltende Schätzung!
In dem oben genannten Video weist Sydney Brenner auch auf ein weit verbreitetes Missverständnis hin, was den Begriff junk DNA angeht. Mit junk wird nämlich im Englischen keineswegs der Müll gemeint, den man wegwirft. Eine bessere Übersetzung wäre wohl das Wort Gerümpel; laut Brenner Dinge, die man gerade nicht brauchen kann, die man deshalb auf dem Dachboden lagert.
Most of the genome appears to be unneccesary; it is - if you like - call it rubbish. Now as you well know, there are two kinds of rubbish: The rubbish you keep, which we call junk, and the rubbish we throw away, which we call garbage.
Dieses Verständnis von junk DNA löst dann auch eine häufig gegen sie gerichtete Kritik in Wohlgefallen auf: Nur weil aktuell kein Nutzen für diese Elemente vorhanden ist, heißt das noch lange nicht, dass diese Sequenzen nicht irgendwann mal benutzt werden können!
[1] von der aktuellen Diskussion über stochastische Transkription und dem Nutzen der so hergestellten RNA mal abgesehen.